이인석 연세대 교수 연구팀 '웹기반 예측 시스템 머핀' 개발
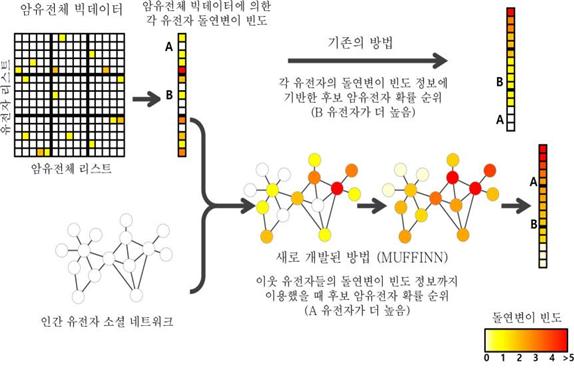
이인석 연세대 교수 연구팀은 암 유전체 빅데이터에서 인간의 유전자들 사이의 기능적 관계를 네트워크로 보여주는 '유전자 소셜 네트워크'를 이용해 암 유전자를 찾아낼 수 있는 '웹기반 예측 시스템 머핀(MUFFINN)'을 개발했다고 26일 밝혔다.
암 진단과 치료를 위해서는 유전체 빅데이터를 생성하고 유전체 염기서열을 정상조직 유전체의 염기서열과 비교·분석하게 된다. 이후 돌연변이를 많이 보이는 유전자를 암 유전자로 발굴하는 것이 현재 발굴 방법이다.
하지만 이는 돌연변이의 빈도가 낮은 암 유전자는 예측이 불가능하다는 한계가 있었다.
연구팀은 암이 단일 유전자 손상에 의한 질환이 아니라 관련 유전자 네트워크의 손상에 의한 질환이라는 암 유전자 네트워크 가설에 근거해 연구를 진행했다.
즉 이웃하는 유전자들의 돌연변이 정보도 이용해 자신의 돌연변이 빈도가 낮아도 이웃 유전자의 돌연변이 빈도가 높으면 암 유전자로 예측하는 방법이다.
연구팀에 의하면 개발된 방법을 기존 암 유전체 빅데이터와 통합해 분석하면 현재 알려진 3~400개의 암 유전자보다 더 많은 새로운 암 유전자 발굴이 가능하게 된다.
실제 국제 유전체연구팀(TCGA)이 발표한 18종의 암 유전체 빅데이터를 개발된 시스템에 적용, 알려진 암 유전자 뿐만 아니라 새로운 암 유전자도 효과적으로 예측할 수 있었다는 게 연구팀의 설명이다.
이인석 교수는 "이번 연구는 그동안 돌연변이 빈도가 낮아서 발굴이 어려웠던 암 유전자들도 찾아낼 수 있는 시스템을 개발한 것"이라면서 "앞으로 환자 수가 적은 희귀암 등 암 유전체 연구에 기여할 것으로 기대된다"고 연구의의를 설명했다.
이번 연구는 미래창조과학부 기초연구사업(개인연구), 바이오의료기술개발사업의 지원으로 연구를 수행됐다. 결과는 게놈 바이올로지(Genome Biology) 23일자에 게재됐다.
댓글 정렬